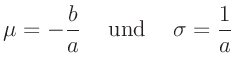![\begin{figure}\begin{picture}(65,65)
\put(0,0){
\frame{
%% 65 x 65 mm
\inclu...
...raphics[width=65mm]{Abbildungen/F1-Abbildung-1b} } }
\end{picture}
\end{figure}](Timg1.png) |
Der Versuch F1 (Einführungspraktikum (2007) Seite 1 und 2) beschäftigt sich mit einer Ursache der Messunsicherheit und deren statistischen Verteilung. Messen bedeutet Vergleichen. Eine physikalische Größe, z.b. eine Länge, wird mit Hilfe eines Messgerätes, z.b mit Hilfe eines Lineals, mit einem definierten Normal verglichen. Um diesen Vergleich für unterschiedliche Werte der zu messenden Größe zu vereinfachen, ist das Messgerät fast immer mit einer Skala ausgestattet, auf der sich der gesuchte Wert ablesen lässt, entweder direkt oder mit Hilfe eines Zeigers. Der Ablesevorgang selbst ist eine Ursache der Messunsicherheit, unabhängig davon, ob der Experimentator diesen selbst ausführt oder die Umsetzung mit elektronischen Hilfsmitteln (digitale Messgeräte) erfolgt.
Den Versuch F1 kann jeder auf seinem eigenen Computer durchführen. Dazu
werden das Programm
GnuPlot
und das GnuPlot-Skript
F1.gnuplot
benötigt. Durch Eingabe des Befehls
load "F1.gnuplot"
in das Terminalfenster von GnuPlot wird der Versuch gestartet.
In dem sich öffnenden Grafikfenster wird nach einem Vorspann eine Skala von 0 bis 10 mit einer Unterteilung in Schritten von 0.5 gezeigt. An einer zufällig gewählten Position dieser Skala ist ein Zeiger dargestellt (linke Seite von Abbildung 1). Die Position des Zeigers soll mit 2 Nachkommastellen geschätzt und notiert werden. Nach 5 Sekunden wird darunter der wahre Wert der Zeigerposition, auf drei Nachkommastellen gerundet, für weitere 4 Sekunden angezeigt (rechte Seite von Abbildung 1). Dieser ist ebenfalls zu notieren. Auf diese Weise entsteht eine Messwertetabelle mit 100 Wertepaaren.
Die weitere computergestützte Auswertung kann auch mit dem Programm
GnuPlot erfolgen. Dazu ist als erster Schritt diese Tabelle mit
einem einfachen Texteditor in eine Datei, z.B. ,,daten.txt``
zu übertragen, jedes Wertepaar in eine eigene Zeile, in der ersten
Spalte der abgelesene Wert, in der zweiten Spalte der wahre Wert.
Die Differenz zwischen abgelesenem Wert und wahrem Wert ist der
Ablesefehler, dessen Statistik im weiteren untersucht werden soll.
Gibt man in das Terminalfenster von GnuPlot den Befehl
stats "data.txt" using ($1-$2);
so erhält man eine Vielzahl von Informationen über die Verteilung des
Ablesefehlers. Sollte stattdessen die Warnung ``Can't read data
file'' erscheinen, ist mit dem Befehl
cd "Pfad zur Datei data.txt"
in das Verzeichnis zu wechseln, in dem die Datendatei gespeichert ist.
Der aktuelle Pfad, das aktuelle Arbeitsverzeichnis, kann mit
pwd
abgefragt werden. Mit den Pfeiltasten up und down kann in
der Liste der zuvor eingegebenen Befehle geblättert werden. Damit
können diese erneut editiert und ausgeführt werden.
Für die weitere Auswertung werden die Größen ![]() ,
, ![]() und
die Anzahl der Datenpunkte benötigt.
und
die Anzahl der Datenpunkte benötigt.
print STATS_min
print STATS_max
ntotal = STATS_records
Für die Darstellung des Histogramms sollten die Werte für ![]() und
und
![]() von Hand so festgelegt werden, dass alle Datenpunkte
enthalten sind. In diesem Beispiel:
von Hand so festgelegt werden, dass alle Datenpunkte
enthalten sind. In diesem Beispiel:
xmin = -0.26
xmax = 0.26
Für die spätere Verwendung können auch gleich die beiden Werte für den
Mittelwert und die Standardabweichung übernommen werden.
mw = STATS_mean
std = STATS_stddev
Zur Darstellung der Häufigkeitsverteilung wird der Bereich
![]() in eine geeignete Anzahl von gleich großen
Intervallen unterteilt. In jedem sollte wenigstens 1 Messpunkt
enthalten sein. Für die Anzahl der Intervalle gibt es unterschiedliche
Empfehlungen. Im Einführungsskript (2007) wird auf Seite 23 ohne
weitere Begründung
in eine geeignete Anzahl von gleich großen
Intervallen unterteilt. In jedem sollte wenigstens 1 Messpunkt
enthalten sein. Für die Anzahl der Intervalle gibt es unterschiedliche
Empfehlungen. Im Einführungsskript (2007) wird auf Seite 23 ohne
weitere Begründung
![]() angegeben. Viele
Statistiklehrbücher beziehen sich auf Sturges (1926), der
für die Intervallebreite
angegeben. Viele
Statistiklehrbücher beziehen sich auf Sturges (1926), der
für die Intervallebreite
![]() vorschlägt.
vorschlägt. ![]() ist der Bereich der Datenwerte
ist der Bereich der Datenwerte
![]() .
Scott (1979) nutzt die aus den Daten bestimmte
Standardabweichung um die Intervallbreite festzulegen.
.
Scott (1979) nutzt die aus den Daten bestimmte
Standardabweichung um die Intervallbreite festzulegen.
![]() . Aus der Intervallbreite
und dem Bereich der Datenwerte ergibt sich dann die Intervallanzahl.
Hier wird mit
. Aus der Intervallbreite
und dem Bereich der Datenwerte ergibt sich dann die Intervallanzahl.
Hier wird mit ![]() weitergerechnet.
weitergerechnet.
ncolumn = 10
Als nächster Schritt ist die Häufigkeitstabelle zu erstellen. Diese
enthält in der ersten Spalte den Mittelpunkt
![]() des
jeweiligen Intervalls, in der zweiten Spalte die Anzahl der
Messpunkte
des
jeweiligen Intervalls, in der zweiten Spalte die Anzahl der
Messpunkte ![]() die innerhalb des Intervalls liegen. In der dritten
Spalte wird gleich noch der Wert der Summenhäufigkeit, der Summe
die innerhalb des Intervalls liegen. In der dritten
Spalte wird gleich noch der Wert der Summenhäufigkeit, der Summe
![]() eingetragen. Diese Tabelle, die in einer zweiten
Datei z.B. ,,histogrammdata.txt`` abgespeichert wird,
kann entweder per Hand oder auch mit dem Programm GnuPlot
erstellt werden. Dazu ist als nächstes die Intervallbreite zu
berechnen, der Dateinamen festzulegen und der Startwert für die
Summenhäufigkeit auf 0 zu setzen.
eingetragen. Diese Tabelle, die in einer zweiten
Datei z.B. ,,histogrammdata.txt`` abgespeichert wird,
kann entweder per Hand oder auch mit dem Programm GnuPlot
erstellt werden. Dazu ist als nächstes die Intervallbreite zu
berechnen, der Dateinamen festzulegen und der Startwert für die
Summenhäufigkeit auf 0 zu setzen.
xdelta = (xmax-xmin)/ncolumn
set print 'histogrammdata.txt'
shn = 0
Durch Verwendung des schon bekannten Kommandos stats lässt sich
die Anzahl der in jedem Intervalle enthaltenen Messwerte
abfragen. Dazu ist die Angabe der Bereichsgrenzen notwendig. Da
wir nur eine Spalte von Werten, die Differenz zwischen Messwert
und wahrem Wert, verarbeiten, wird diese von GnuPlot als Spalte
von y-Werten betrachtet. Als x-Werte werden in diesem Fall die
Zeilennummer in der Datendatei angesehen.
Mit dem komplexen Befehl
do for [i = 1:ncolumn] {
xmax = xmin + xdelta
set yrange [xmin:xmax]
stats "data.txt" using ($1-$2) nooutput;
n = STATS_records
shn = shn + n
print sprintf("% .3f %3i %3i",(xmin+xmax)/2, n, shn)
xmin = xmax
}
wird die Datei mit den Histogrammdaten erzeugt. Erst nach Eingabe der
letzten schließenden Klammer erfolgt die Bearbeitung. Die
Fehlermeldung ,,All points out of range`` bedeutet, dass ein
Intervall keine Datenpunkte enthält. In diesem Fall ist die Anzahl der
Intervalle zu verringern oder die Werte von ![]() und
und
![]() leicht zu verändern. Als nächstes ist die Datei mit den
berechneten Histogrammdaten noch mit dem Kommando
leicht zu verändern. Als nächstes ist die Datei mit den
berechneten Histogrammdaten noch mit dem Kommando
set print
zu schließen. Der letzte Wert der Summenhäufigkeit sollte mit der
Anzahl der Datenpunkte übereinstimmen. Dies lässt sich leicht mit
print shn, ntotal
überprüfen. Die Festlegung der y-Bereichsgrenzen ist auf das
Standardverhalten zurückzusetzen
unset yrange
und die Achsenbeschriftung festzulegen
set xlabel "Messabweichung"
set ylabel "absolute Häufigkeit"
Nun kann das Histogramm gezeichnet werden.
plot "histogrammdata.txt" using 1:2 with boxes notitle;
Die Höhe der einzelnen Balken gibt die Anzahl der Datenpunkte in den
jeweiligen Intervall an. Das Verhältnis
![]() entspricht der
Wahrscheinlichkeitsdichtefunktion der zu Grunde liegenden
statistischen Verteilung, die sich für
entspricht der
Wahrscheinlichkeitsdichtefunktion der zu Grunde liegenden
statistischen Verteilung, die sich für
![]() und
und
![]() ergäbe. In
sehr vielen Fällen ist dies die Dichtefunktion der Gauß-Verteilung
ergäbe. In
sehr vielen Fällen ist dies die Dichtefunktion der Gauß-Verteilung
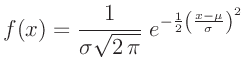
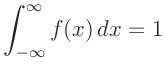
Wesentlich besser lässt sich die Übereinstimmung zwischen der Verteilung der Datenpunkte und einer gegebenen Wahrscheinlichkeitsverteilung mit Hilfe der Verteilungsfunktion beurteilen. Diese ist allgemein definiert durch:
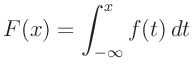
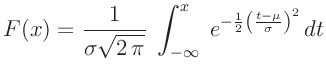
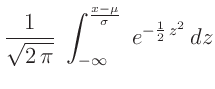 |
|||
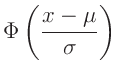 |
Die Abweichungen sind bei dieser Darstellung (Abbildung 3) besser zu erkennen. Noch deutlicher könnten diese werden, wenn die y-Achse des Graphen in einer solchen Weise geteilt wäre, dass die Verteilungsfunktion eine Gerade ergäbe. Dies ist durch eine Skalierung mit der Umkehrfunktion der Verteilungsfunktion (Gleichung 1) zu erreichen.
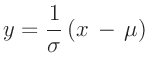
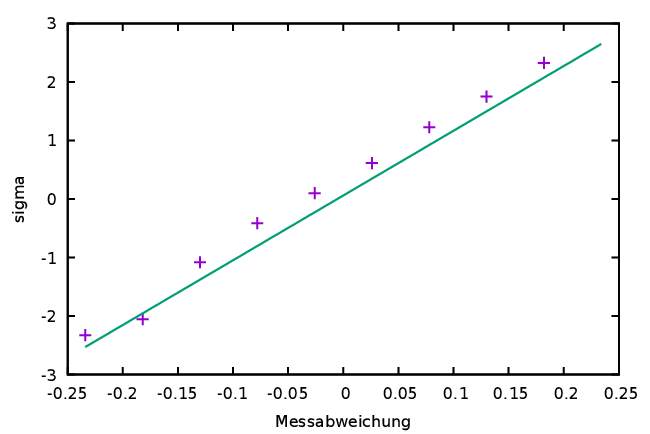 |
Bei dieser Darstellung (Abbildung 4) ist
die y-Achse in Vielfachen von ![]() geteilt.
Die Werte der Summenhäufigkeit sind nicht mehr direkt ablesbar. Um das
zu ändern, kann die rechte y-Achse mit einer entsprechend skalierten
Einteilung versehen werden. Dazu sind die folgenden Befehle notwendig,
geteilt.
Die Werte der Summenhäufigkeit sind nicht mehr direkt ablesbar. Um das
zu ändern, kann die rechte y-Achse mit einer entsprechend skalierten
Einteilung versehen werden. Dazu sind die folgenden Befehle notwendig,
set yrange [-3.0:3.0]
set y2range [0:1.0]
set ytics nomirror
set link y2 via norm(y) inverse invnorm(y)
set y2tics 0.1 format "%.2f" nomirror
set y2tics add (0.01, 0.05, 0.95, 0.99)
mit denen die Skalierung der y2-Achse über die Verteilungsfunktion mit
der y-Achse verknüpft wird. Zusätzlich sollten beide Achsen
beschriftet werden.
set ylabel "sigma"
set y2label "relative Summenhäufigkeit"
Um die Ablesbarkeit der Werte aus dem Graphen zu erleichtern lässt
sich mit
set grid xtics y2tics
noch ein Koordinatennetz hinzufügen. Dieses Netz, auf Papier
ausgedruckt, wurde, als ,,Wahrscheinlichkeitspapier``
bezeichnet, in Vor-Computer-Zeiten für den schnellen Vergleich einer
empirisch bestimmten Verteilung mit der Normalverteilung
verwendet. Nun kann alles zusammen noch einmal neu gezeichnet werden.
plot "histogrammdata.txt" using 1:(invnorm($3/ntotal)) notitle,\
((x-mw)/std) with lines notitle;
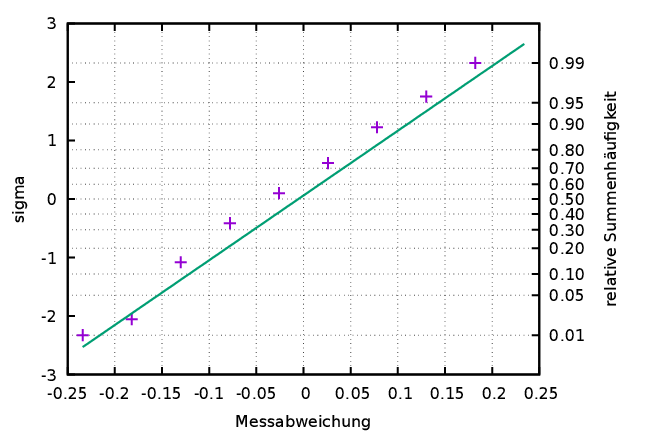 |
Die in den Abbildung 4 und
5 eingezeichnete Gerade wurde auf
Grundlage der am Anfang abgeschätzten Werte für ![]() und
und ![]() berechnet. Es stellt sich nun die Frage, ob es nicht eine besser an
die Datenpunkte angepasste Gerade gibt. Dazu kann wieder der Befehl
stats verwendet werden. Nur wird er jetzt auf die
Histogramm-Daten, auf die Spalten mit
berechnet. Es stellt sich nun die Frage, ob es nicht eine besser an
die Datenpunkte angepasste Gerade gibt. Dazu kann wieder der Befehl
stats verwendet werden. Nur wird er jetzt auf die
Histogramm-Daten, auf die Spalten mit
![]() und
Summenhäufigkeit angewandt. Die Summenhäufigkeiten werden dabei mit
der inversen Verteilungsfunktion (Gleichung 2) skaliert.
und
Summenhäufigkeit angewandt. Die Summenhäufigkeiten werden dabei mit
der inversen Verteilungsfunktion (Gleichung 2) skaliert.
stats "histogrammdata.txt" using 1:(invnorm($3/ntotal));
Als Ergebnis erhält man neben den statistischen Informationen zu den
Daten in den beiden Spalten auch Informationen zu den statistischen
Beziehungen zwischen den beiden Datenspalten. Darunter auch die Parameter
![]() und
und ![]() der Geraden
der Geraden
![]() , die einen vorhandenen linearen
Zusammenhang am besten beschreibt. Diese können mit den Befehlen
, die einen vorhandenen linearen
Zusammenhang am besten beschreibt. Diese können mit den Befehlen
a=STATS_slope
b=STATS_intercept
übernommen werden. Die Graphik wird mit der angepassten Geraden
y(x)=a*x+b
erneut gezeichnet (Abbildung 6).
plot "histogrammdata.txt" using 1:(invnorm($3/ntotal))
notitle, \
y(x) with lines notitle;
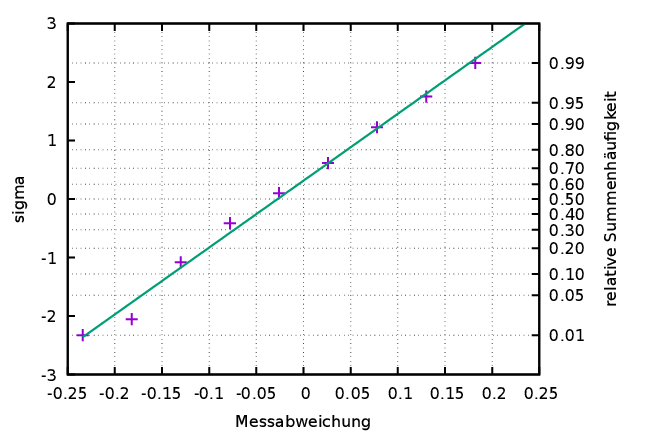 |